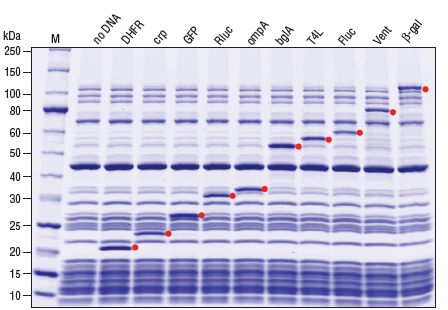
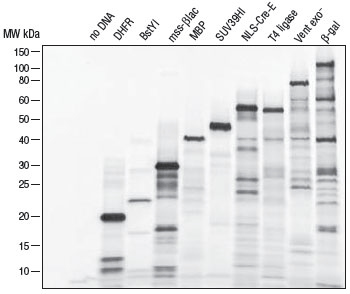
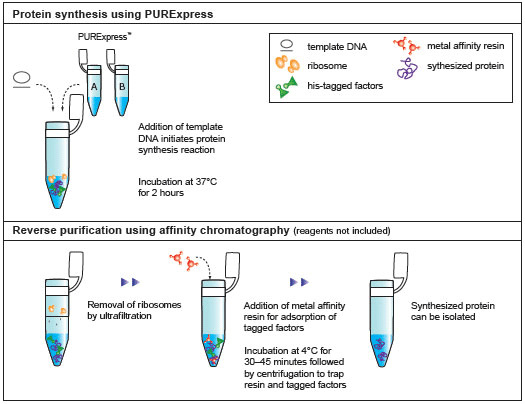
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
产品信息

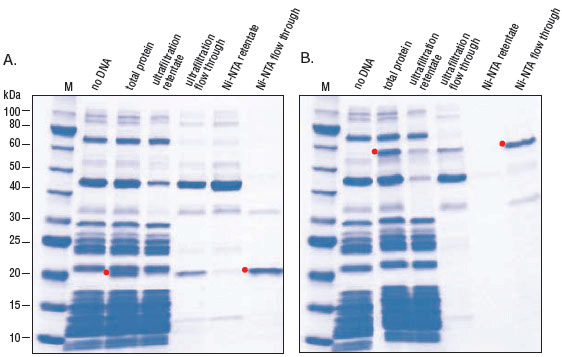
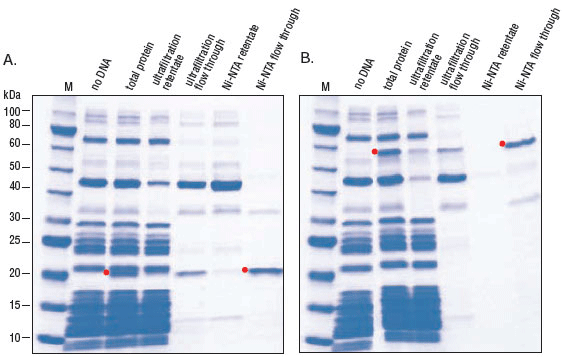
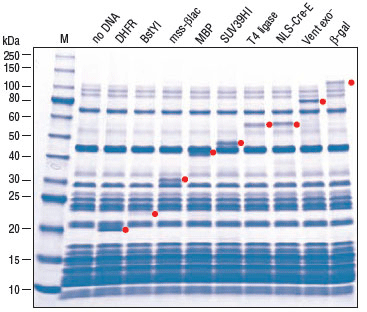
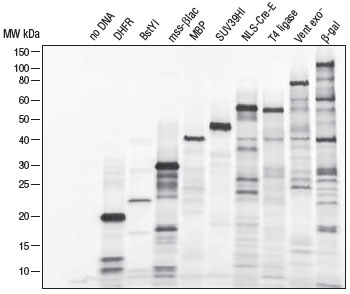
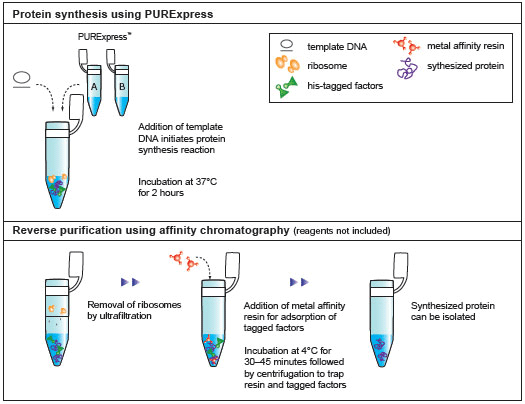
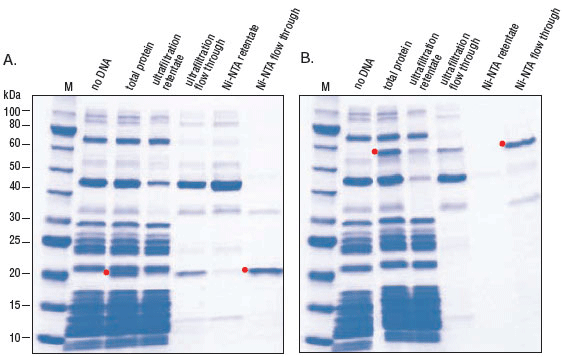
(A.)反应按照 PURExpress 体系说明建立,以vtPA DNA 为模板。37℃温育 2 小时后,取 5 μl 反应液进行活性检测,在 1 小时内监测发光底物的剪切反应。(B.)各取 2.5 µl 反应液,用 SDS-PAGE 电泳分离,使用考马斯亮蓝染色。vtPA 目的蛋白用红色箭头标记。
添加 PDBE 产生活性蛋白。通过对靶标的功能活性检测,发现活性差异是由二硫键的折叠所致,而不是由于 PURExpress 体系合成蛋白产量导致的差异。


- 产品类别:
- NEBExpress® Cell-free E. coli Protein Synthesis System,
- PURExpress,
- Cell-Free Protein Expression Products,
Protein Expression Products
- 应用:
- NEBExpress® Cell-free E. coli Protein Synthesis System,
- PURExpress,
- Cell-Free Protein Expression,
- Expression of Difficult Proteins,
- Disulfide-bonded Protein Expression,
Protein Expression
-
试剂盒组成
本产品提供以下试剂或组分:
NEB # 名称 组分货号 储存温度 数量 浓度 -
E6820S -20 PURExpress Disulfide Bond Enhancer 1 P6821AVIAL -20 1 x 0.05 ml 25 X Enhancer 2 B6822AVIAL -20 1 x 0.05 ml 25 X
-
-
相关产品
相关产品
- PURExpress® 体外蛋白合成试剂盒
- PURExpress® Δ Ribosome 试剂盒
- PURExpress® Δ (aa, tRNA) 试剂盒
- PURExpress® Δ RF123 试剂盒
-
注意事项
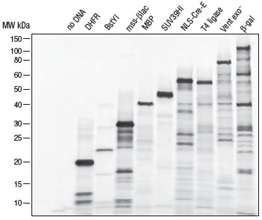
- PURExpress 二硫键增强剂经过优化,能够与 NEB PURExpress 体系(NEB #E6800、E3313)一起使用。然而,它与其它供应商的细菌 S30 裂解物 IVTT 体系兼容(图 2)。由于其它体系的产品剂型不同,因此与 PDBE 相关的所有指南、操作说明和常见问题解答均仅适用于 PURExpress 体系。
-
参考文献
- Harada R, Furumoto S, Yoshikawa T, Ishikawa Y, Shibuya K, Okamura N, Ishiwata K, Iwata R, Yanai K (2016). Synthesis and Characterization of 18F-Interleukin-8 Using a Cell-Free Translation System and 4-18F-Fluoro-L-Proline.. Journal of Nuclear Medicine. 57 (4), 634-639. DOI: 10.2967/jnumed.115.162602 PubMedID: 26742712
- Horiya S, Bailey JK, Krauss IJ (2017). Directed Evolution of Gycopeptides Using mRNA Display.. Methods in Enzymology . DOI: 10.1016/bs.mie.2017.06.029 PubMedID: 28935113
- Huang A, P.Q. Nguyen, J.C. Stark, M.K. Takahashi, N. Donghia, T. Ferrante, A.J. Dy, K.J. Hsu, R.S. Dubner, K. Pardee, M.C. Jewett and J.J. Collins (2018). Biobits™ Explorer: A modular synthetic biology education kit. Science Advances. 4 (8),
- Mankowska, S.A., P. Gatti-Lafranconi, M. Chodorge, S. Sridharan, R.R. Minter and F. Hollfelder (2016). A Shorter Route to Antibody Binders via Quantitative in vitro Bead-Display Screening and Consensus Analysis. Scientific Reports. 6,
- Pardee K, Slomovic S, Nguyen PQ, Lee JW, Donghia N, Burrill D, Ferrante T, McSorley FR, Furuta Y, Vernet A, Lewandowski M, Boddy CN, Joshi NS, Collins JJ (2016). Portable, On-Demand Biomolecular Manufacturing. Cell. 248-259 e212. DOI: 10.1016/j.cell.2016.09.013 PubMedID: 27662092
- Price A.K., MacConnell A.B., Paegel B.M. (2014). Microfluidic bead suspension hopper. Analytical Chemistry. 86 (10), 5039-5044. DOI: 10.1021/ac500693r PubMedID: 24761972
- Price A.K., MacConnell A.B., Paege B.M. (2016). hnuSABR: Photochemical Dose-Response Bead Screening in Droplets.. Analytical Chemistry. 88 (5), 2904-2911.
- Ren G, Ke N, Berkmen M (2016). Use of the SHuffle Strains in Production of Proteins. Current Protocols in Protein Science. 85, DOI: 10.1002/cpps.11 PubMedID: 27479507
- Tran D.T., Cavett V.J., Dang V.Q., Torres H.L., Paegel B.M. (2016). Evolution of a mass spectrometry-grade protease with PTM-directed specificity. Proceedings of the National Acadmey of Sciences of the United States of America. 113 (51), 14686-14691. DOI: 10.1073/pnas.1609925113 PubMedID: 27940920
操作说明、说明书 & 用法
-
操作说明
- PURExpress Disulfide Bond Enhancer (E6820)
-
应用实例
- AppNote_Use_of_PURExpress _and_PDBE_to_Express_Active_Proteins_that_can_be_Expressed_in vivo_in_Shuffle_Competent_Cells
- NEBExpress® Cell-free E. coli Protein Synthesis System
FAQs & 问题解决指南
-
FAQs
- Is the PURExpress® In Vitro Protein Synthesis Kit capable of dealing with disulfide bonds? If not, can you recommend something else to use post synthesis?
-
实验技巧
- 可能需要对溶液 1 和溶液 2 进行滴定测试,以获得最佳的蛋白折叠。每种溶液从 1 ul 的量开始。
可与 S30 裂解物和 PURExpress 一起使用。 - 可能需要对溶液 1 和溶液 2 进行滴定测试,以获得最佳的蛋白折叠。每种溶液从 1 ul 的量开始。