上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
产品信息


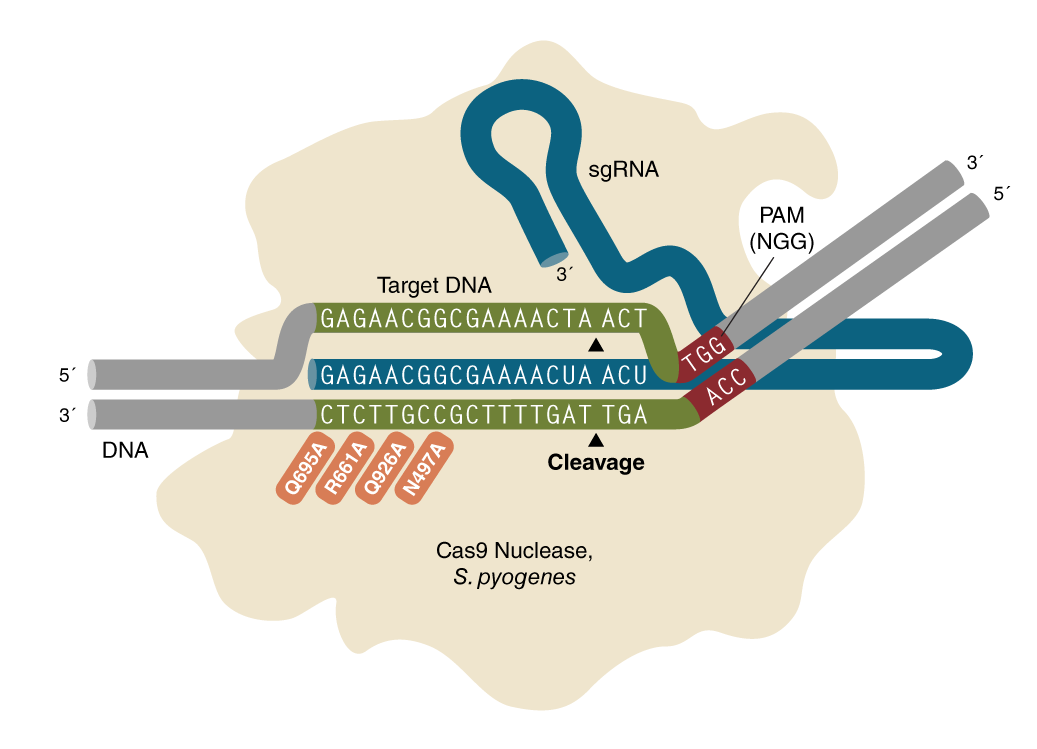
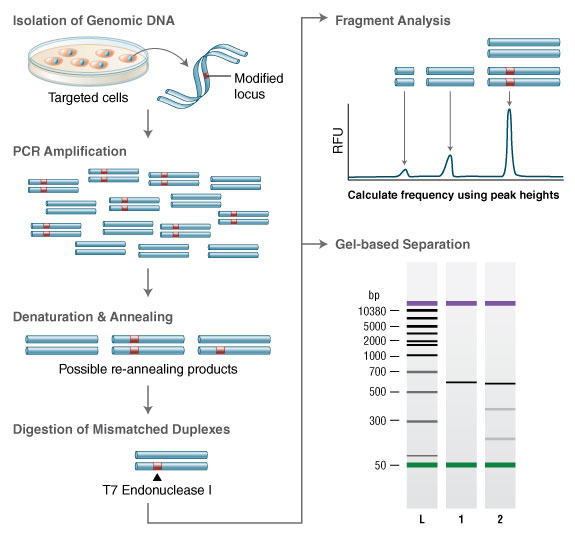
EnGen 突变检测试剂盒为检测基因组靶标编辑效率提供了一整套试剂。第一步,使用 Q5 热启动超保真 2X 预混液从待测细胞中扩增出目的基因组的靶标区域(即通过 CRISPR/Cas9、TALEN 或锌指核酸酶等技术进行了基因编辑的区域)。经变性和重新退火后,当扩增子池中出现基因插入和缺失(InDel)等造成的突变时,就会形成异源双链 DNA。第二步,将退火后的 PCR 产物用 EnGen T7 核酸内切酶 I 进行酶切。EnGen T7 核酸内切酶 I 是一种结构特异性酶,可有效识别大于 1 个碱基的基因错配。当错配出现时,DNA 的两条链都会被切割形成较小的片段。分析片段图谱可以对基因组编辑实验的效率进行评估。
EnGen 突变检测试剂盒包含一组对照模板和引物预混液,可用作 PCR 反应及 T7 核酸内切酶 I 酶切实验的对照。对照模板和引物预混液中包含两种对照质粒和一对对照引物,经扩增、变性、重新退火后,会形成含有 10 个碱基插入的异源双链 DNA。此种异源双链 DNA 正是 T7 核酸内切酶 I 的底物。包含扩增子的 600 bp 异源双链 DNA 会被切割为 200 bp 和 400 bp 两个片段;而 600 bp 同源双链 DNA 则不会被 T7 核酸内切酶 I 切割。通过琼脂糖凝胶电泳或片段分析仪器可以轻松分辨出同源双链与经切割的异源双链片段。
试剂盒的实验流程经过优化,通过 Q5 热启动超保真 2X 预混液扩增得到的 PCR 产物可以不经纯化而直接用 T7 核酸内切酶 I 进行消化。异源双链 DNA 只需 15 分钟就可以被完全酶切,随后用蛋白酶 K 终止反应。此外,Q5 热启动超保真 2X 预混液还可在酶切前用来优化靶点扩增。
图 1:突变检测试剂盒实验流程
- 产品类别:
- Mutation Detection Reagents and Kits Products,
- Genome Editing Products,
- DNA Repair Enzymes and Structure-specific Endonucleases Products
- 应用:
- Genome Editing Applications
-
试剂盒组成
本产品提供以下试剂或组分:
NEB # 名称 组分货号 储存温度 数量 浓度 -
E3321S -20 Q5® Hot Start High-Fidelity 2X Master Mix M0494AAVIAL -20 1 x 0.4 ml 2 X NEBuffer™ 2 B7002SVIAL -20 1 x 1.25 ml 10 X Control Template and Primer Mix S0534AVIAL -20 1 x 0.025 ml 10 X Proteinase K, Molecular Biology Grade P8107AVIAL -20 1 x 0.12 ml 800 units/ml Quick-Load® Purple 1 kb Plus DNA Ladder N0550AVIAL 4 1 x 0.2 ml 100 µg/ml Gel Loading Dye, Purple (6X), no SDS B7025SVIAL 25 1 x 1 ml 6 X EnGen® T7 Endonuclease I M0450AVIAL -20 1 x 0.025 ml Not Applicable
-
-
特性和用法
需要但不提供的材料
用于 PCR 的寡脱氧核苷酸引物
PCR 管或联排管
无核酶水
热循环仪
用于 PCR 的防气溶胶吸头 -
相关产品
相关产品
- Spy Cas9 核酸酶
- 6X 紫色凝胶上样染料,无 SDS
- Quick-Load® 紫色 1 Kb Plus DNA Ladder
- Q5® 热启动超保真 2X 预混液
- Monarch® PCR & DNA 纯化试剂盒(5 μg)
- e2050-hiscribe-t7-quick-high-yield-rna-synthesis-kit
- NEBuilder® 高保真 DNA 组装克隆试剂盒
- NEBuilder® 高保真 DNA 组装预混液
- EnGen® sgRNA 合成试剂盒,S. pyogenes
操作说明、说明书 & 用法
-
操作说明
- T7 Endonuclease I-based Mutation Detection with the EnGen® Mutation Detection Kit (NEB #E3321)
- Transfection of Cas9 RNP (ribonucleoprotein) into adherent cells using the Lipofectamine® RNAiMAX
-
说明书
产品说明书包含产品使用的详细信息、产品配方和质控分析。- manualE3321
-
应用实例
- Determining Efficiency of On-Target CRISPR Cas9 Genome Editing Using the NEB® EnGen® Mutation Detection Kit on LabChip Gel Xpress
FAQs & 问题解决指南
-
FAQs
- Why do I see an extra band when I run the undigested heteroduplex on an agarose gel?
- Why do I see very little DNA on my gel or low signal on the fragment analyzer?
- Will EnGen® T7 Endonuclease I recognize single base mismatches and single base insertions or deletions (indels)?
- Can cell lysates be used in the PCR reaction?
- What are the expected results using the included control?
- My PCR reaction yield is low, can I add more than 5 µl of the PCR reaction to the digestion reaction?
-
问题解决指南
- PCR Troubleshooting Guide