Q&A
Q: DsDD cDNA Subtraction Kit做什么的试剂盒?
A: 将需要做比较的cDNA库用PCR进行扩增制备Tester和Driver cDNA,对DNA扩增产物进行削减的试剂盒。与传统方法相比,操作更简便,实
验时间也仅需2天时间。可以将现有的cDNA库或市面售卖的cDNA库直接开始进行实验,这是传统方法所不能做到的。而且最终制备出来的
Subtraction cDNA是混入管家基因较少的cDNA,可以直接应用于各种用途。
Q: DsDD cDNA Subtraction Kit有什么优点?
A:● 直接使用现有的cDNA,全部工序完成仅需2天。
A:● 全程在DNA状态下进行,无需熟练的操作技术。cDNA的制备有非常高的选择性。
A:● 可以通过市面或用户自己准备的质粒cDNA来制备。
A:● 除了质粒以外,还可以从5’和3’端带接头的cDNA制备cDNA。
A:● 操作简便
A:● 最终制备出来的Subtraction cDNA反映最初所用的Tester cDNA。因为Tester没有经过限制性内切酶处理,所以一开始的Tester cDNA可
以反映最终制备的Subtraction cDNA的状态。原理上,在载体库使用全长cDNA,就能反映Subtraction cDNA的全长。
A:● 只要改变Tester和Driver,就能进行反向削减。
A:● 制备的Subtraction cDNA可以立刻应用于各种用途。
A:● 制备的Subtraction cDNA中,混入管家基因的量少,所以它还可以作为载体亚克隆(系统序列)和DNA芯片的探针使用。
Q: 与PCR-Select法有什么不同?
A:
|
起始材料
|
接头添加
|
杂交次数
|
house-keeping基因污染
|
|
DsDD法
|
cDNA库或5’和3’末端带接头的cDNA
|
不必要
|
1次
|
已经分解去除,污染小
|
|
PCR-Select法
|
RNA
|
必要
|
2次
|
未经分解去除,有污染的可能性
|
● PCR-Select法每次都需要通过RNA制备cDNA,DsDD法则只需cDNA库就能进行实验。另外,采用PCR-Select法必须用Rsal(4碱基内切酶)
酶切,将低分子化的接头分子与3’或5’端结合。而DsDD不需要。因此最终的Subtraction cDNA能反映最初的Tester cDNA的状态。
● PCR-Select法需要进行两次杂交,而DsDD法只需一次。
● PCR-Select法需要从大量cDNA样品中用Supression-PCR对SubtactedcDNA进行特异性扩增,而DsDD法则是通过对与管家基因等共同表达
的基因进行杂交后,用分解去除的方式,极力减少管家基因的污染。DsDD法的原理上是形成Subtraction cDNA群,因此可以直接应用于克
隆和DNA芯片的制作。
Q: 操作DsDD法时,特别需要注意的事项是什么?
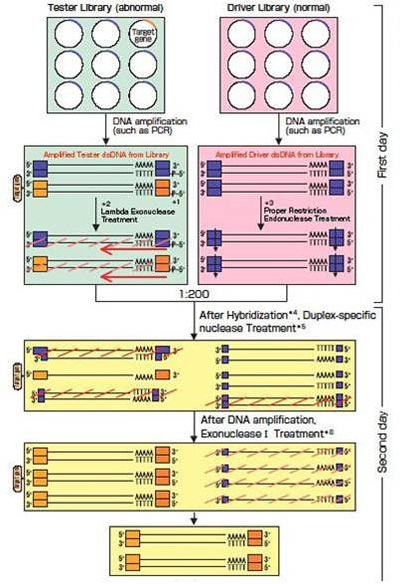
A:1. 使用Tester,运用单向插入的cDNA库。
Tester的cDNA库,必须对载体使用单向插入的cDNA库。若不用单向cDNA库,即使是用λ核酸外切酶处理成单链,有意链和反意链的
cDNA会混为一体,Tester聚集杂交,是降低Subtraction效率的主要原因。
A:2. 使用去除低分子DNA的色谱柱
DSN(Duplex-specific nuclease)是8bp以上的完全一致的双链DNA分解酶,因此其短断片残留多。在乙醇沉淀中,无法去除这些断片
或引物。引物残留的非特异性杂交,是DSN的非特异性分解的原因。DSN的分解产物在最后的PCR时,变成非特异性引物,是形成低分
子断片等非特异性断片增加的原因。
A:3. 用乙醇共沉淀时,务必使用配套的Ethachinmate(乙醇沉淀核酸载体)
共沉淀时使用tRNA等核酸,会与Tester cDNA发生非特异性杂交。引起DSN的非特异性分解。
而Ethachinmate(乙醇沉淀核酸载体)是聚丙烯酰胺类的共沉淀剂,DNA回收率高达100%。即使通常不可见的沉淀也可以变为可见,
在操作移液器时避免误吸。
A:4. 使用适用于1 μL的移液器。
A :1 μL的移液操作时,尽可能使用合适的吸移管操作(如Gilson公司的PIPETMAN 2P)。特别是在进行杂交时,Tester ssDNA和Driver
dsDNA需要注意移液操作。这一操作中,Tester:Driver=1:200这一比率的把握尤为重要,尽可能减小在移液移过程中的误差。
A:5. 酶反应时使用PCR管。
说明书记载DNA扩增反应应尽可能使用PCR管。PCR热效率较好可以进行正确的反应。
Q: 除试剂盒外还需准备什么?
A:1. Tester和Driver扩增用的cDNA库
A:2. Tester和Driver扩增用的Primer
A:3. 参照基因扩增用Primer(GAPDH、β-Actin等)
A:4. DNA聚合酶(TOPOTAQ DNA polymerase等)
A:5. dNTP Mixture
A:6. 低分子DNA和Primer去除用色谱柱
A:7. 限定性酶
A:8. 苯酚:氯仿:异戊醇(25:24:1)
A:9. 70%、99.5%乙醇
A:10. DNA分子量marker
A:11. 琼脂糖
A:12. 溴化乙锭
A:13. 1×TAE Buffer
A:14. Loading Buffer
A:15. 灭菌蒸馏水
Q: DsDD是什么的缩写?
A: DsDD是Duplex-specific Direct Digestion(双链特异性直接消化)的缩写。通过Duplex-specific DNA Nucelase,可以特异性切断杂交形
成的DNA群。
Q: Tester和Driver是什么?
A: 特异性表达的cDNA端叫“Tester”,作为基准的cDNA一般称为“Driver”。从癌特异性cDNA库制备的cDNA为“Tester”,正常细胞
来源的cDNA库配制的cDNA则为“Driver”。
Q: 应选用何种凝胶过滤柱?
A: 可以使用能去除100bp以下的,市面售卖的凝胶过滤柱。Code No.:195-14451 Spin Cleaner
Q: 杂交一定要经过16小时么?
A: 16~20小时都是没有问题的。建议16个小时是因为考虑到从实验当天17点开始进行杂交的话,第二天上午9点就可以进行下一项操作。
Q: 按照protocol制备Driver ds cDNA,但还是不能配出200 ng/μL的浓度,应该如何解决?
A: 浓度比较小的时候,请用乙醇共沉淀法操作。离心管中加入Tester 1 ng、Driver 200 ng后,加入无菌水100 μL,Ethachinmate 1μL,
3mol/L的Sodium Acetate 10 μL,乙醇250 μL,振荡混合后进行离心。然后去除上清,加入150 μL 70%的乙醇离心后,去除上清,干
燥,再加入3 μL的灭菌蒸馏水进行溶解。备用于杂交试验。
杂交操作中Tester:Driver=1:200的比率十分重要。乙醇沉淀导致部分DNA缺失是没有问题的。
Q: DSN (Duplex-specific nuclease)有什么优点?
A: DSN是堪察加蟹肝脏来源的新型双链特异性DNA分解酶,可在双链DNA或DNA-RNA杂交过程中特异性切断DNA。最适宜温度55~65℃,
Mg2+条件下,可用EDTA抑制其活性。因为DSN能在高温环境下进行反应,所以在DsDD法中,进行严格性较高的杂交反应。
Q: 一定要使用PCR管吗?
A: 说明书记载DNA扩增反应应尽可能使用PCR管。热效率较好能进行正确的反应。
Q: Drive ds cDNA的限定性酶处理是必要的么?
A: Tester和Driver的载体相同时,需要进行限定性酶处理。目的是防止因Primer结合引起非特异性杂交而导致DSN分解。用限定性酶只剪切
Tester和Driver的cDNA,相辅的cDNA杂交。通过改变Tester和Driver端的Primer set,进而减少非特异性杂交。
Q: 扩增的Tester和Driver侧的Primer set可以是相同的吗?
A: 同一Set也可以,但不是酶处理本身100%的反应。未处理的Driver cDNA Primer和Tester sscDNA的Primer结合,会通过DSN被分解。
为了防止上述情况,建议扩增Tester和Driver端的PrimerSet还是要区分开。
Q: 限定性酶处理时如果出现Driver cDNA的内部被切断的话,会有影响吗?
A: 没有影响。即使被切断了,还是能与Tester sscDNA进行杂交。
Q: 一定要进行Exonuclease I处理吗?
A: 不是必要的操作。对Subtraction cDNA使用标记探针时,Exonuclease I能分解Driver的cDNA,可降低背景。
Q: 最后,扩增削减cDNA用到的Primer需要进行磷酸化吗?
A: 不用,而是需要合成新的Primer。可以的话,建议使用最初的Primer内侧区域的Primer进行PCR操作。
Q: 配制Tester cDNA和Driver cDNA时使用的plasmid DNA会有影响吗?
A: 没有影响的。DsDD法用的是双链特异性DNA分解酶(DSN),可分解plasmid DNA,所以不会影响。像生物素结合法、乳胶微粒oligo
(dT)固相法这些传统方法,无法去除plasmid DNA,才会对转换和探针的制备等产生影响。
Q: 说明书推荐的TOPOTAQ DNA polymerase有什么特点?
A: 本产品是Pyrococcus DNA polymerase和Taq DNA polymerase来源的催化结构域和甲烷细菌Methanopyruskandleri来源的非特异性
DNA结合域融合的新型耐热性DNA Polymarase。本酶家族带有拓朴异构酶活性。通过这个特性,仅通过酶反应就能扩增GC-rich领域。
这是目前其他酶都无法轻易做到的。TOPOTAQ DNA polymerase有热启动功能,能抑制非特异性的扩增。
Q: Ethachinmate是什么?
A: Ethachinmate由NipponGene生产的,在核酸(DNA或RNA)被乙醇或异丙醇沉淀时使用的高分子载体。使用本品,能从浓度低的核酸
溶液中定量回收微量核酸。加入乙醇或异丙醇后,无需进行-20℃或-80℃冷却,可直接离心。回收的核酸用缓冲液溶解后,可直接作为各
种酶的基质使用。
Q: 去除的Subtracted cDNA有多大?
A: 和光确认的大小为500~1500bp。最初用的cDNA库和DNA扩增的的延伸时间会影响Subtracted cDNA的大小。
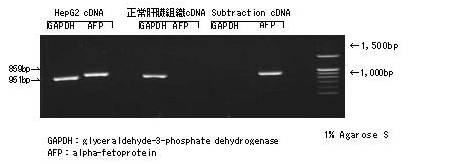
Q: Subtraction的效率是多少?
A: Tester端用肝癌细胞HepG2来源的cDNA,Driver端用人正常肝脏组织由来cDNA的话,Real-time PCR过程中GAPDH会被抑制至1/1,000。
Q: 对削减的基因进行亚克隆化,什么方法比较好?
A: 一般采用的方法是:TA克隆或限定性酶处理后,插入目的载体,进行转换。
Q: 证明cDNA削减的方法是什么?
A: 配制的Subtracted cDNA经过TA克隆后,在菌落PCR确认插入断片,接合探针,通过其在市面售卖的mRNA点膜上的斑点,判断是否有组
织特异性基因存在的。还可以在数据库搜索序列,或解析DNA芯片的方法。
![]()